
Электронные медицинские карты (ЭМК) представляют основу автоматизации медицинской организации. В последнее время они повсеместно внедряются во всем мире.
По некоторым данным около 95% больниц США внедрили самые разнообразные системы ведения ЭМК. У нас в стране благодаря вначале реализации проекта «ЕГИСЗ» в 2011-2018 г., а затем и последовавшему федеральному проекту «Создания единого цифрового контура в сфере здравоохранения на основе ЕГИСЗ» в многих медицинских организация также стали применяться ЭМК.
Некоторые передовые регионы и отдельные медицинские организации уже по 5 и более лет работают с такими системами, накопив поистине огромные базы данных с разнообразными протоколами инструментального и лабораторного обследования, врачебными осмотрами и т.д.
В целом, в отрасли информатизации здравоохранения имеется мнение, что ЭМК редко могут быть использованы для машинной обработки, в особенности для создания на их основе наборов данных для последующего машинного обучения.
Главная причина- преимущественно неструктурированный способ хранения медицинских записей, в особенности врачебных осмотров. И действительно, сегодня большинство систем ведения ЭМК ставят во главу угла удобство пользователей и скорость внесения ими своих записей, что чаще всего делается через разные шаблоны и упрощенные текстовые формы наподобие мини-текстовых редакторов. Причина этого кроется в том, что если заставлять врача вносить информацию через «жесткие» формализованные формы ввода – то такая информация будет более структурированная (и отчасти более качественная), но потребует существенно больше времени на ее внесение.
По наблюдению наших экспертов, порой такой способ требует в 2-3 раза больше времени, чем быстрая коррекция обычного текстового шаблона, подгруженная из справочника текстовых заготовок.

Мы считаем, что такая ситуация будет сохраняться еще достаточно длительное время. Поэтому именно неструктурированные протоколы врачебных осмотров будут превалировать над детальными машинно-читаемыми формализованными клиническими протоколами. Отсюда возникает идея не отрицать неструктурированные медицинские записи, а учиться с ними работать.
Для таких задач создаются специализированные сервисы. Одним из таких решений является Amazon Comprehend Medical, который позволяет извлекать значимую информацию (жалобы, диагноз, назначенные препараты и их дозировку, результаты исследований и т.п) из неструктурированных медицинских записей.
При этом от пользователя не требуется знаний машинного обучения, т.к. Comprehend Medical предоставляется по сервисной модели через интеграционные API Amazon. Этот продукт ориентирован на поставщиков медицинских услуг, страховщиков и исследователей, а также на медицинские, биотехнологические и фармацевтические компании, которым сервис позволит быстро внедрять системы поддержки принятия решений и улучшить процессы управление медицинскими данными пациентов https://aws.amazon.com/ru/blogs/machine-learning/introducing-medical-language-processing-with-amazon-comprehend-medical/ .
Данный подход находит в последнее время подтверждение и в серьезной научной литературе. Например, в конце 2019 г. JAMIA опубликовала исследование, которое показало, что данные, полученные из неструктурированных ЭМК, являются более точным источником информации для прогнозирования ИБС, чем структурированные данные. Это еще больше подкрепляет нашу убеждённость перспектив извлечения признаков из неструктурированных ЭМК, в т.ч. для задач сбора больших наборов данных для машинного обучения. Более того, развитие ИИ позволяет не просто извлекать признаки из текстовых записей – но и демонстрирует, что работа с непосредственными неструктурированными записями также может давать моделям ИИ точность даже выше, чем если это были формализованные признаки https://www.healthcareitnews.com/news/unstructured-ehr-data-more-useful-predictive-analytics-study-shows.
В современных ЭМК до 70% информации написано на естественном языке. Свободный текст удобен для выражения клинических концепций и событий, таких как диагностика, симптомы и вмешательства. Врачи в своих записях фиксируют жалобы пациентов, симптомы и назначенную лекарственную терапию в формате неструктурированных текстов. Многие важные наблюдения остаются незарегистрированными в полях форм протоколов, очень часто можно видеть комментарии врача в виде свободного текста, хранящегося рядом с пустыми полями форм.
Для того, чтобы система «Webiomed» могла «извлекать» из медицинских текстовых записей структурированные признаки, мы создали специальный сервис – Webiomed.NLP.
Для его развития мы создаем модели машинного обучении с использованием методов Natural language processing (NLP). Эти возможности позволят нашей системе из обычных текстовых медицинских протоколов вытаскивать клинически-значимую неразмеченную информацию, которая затем используется для машинного анализа поступивших в Webiomed запросов от медицинских информационных систем.
С помощью NLP мы извлекает симптомы из жалоб, данные об артериальном давлении, росте и весе пациента из неструктурированных объективных данных, лабораторные показатели из выписок и многое другое. Мы также используем Webiomed.NLP для обогащения накапливаемых «сырых данных» (raw data) и создания с помощью этого собственных размеченных дата-сетов, которые нужны нам для последующего машинного обучения и создания прогностических моделей.
Используемые методы
Для анализа медицинских текстовых записей используются специализированные методы и инструменты распознавания именованных объектов Named Entity Recognition (NER). Эти методы применяются для разметки и классификации именованных в тексте частей - классов. Количество классов является заранее известным. Классами могут являться наименования заболеваний , факты летального случая или госпитализации пациента (есть или нет), различные количественные и качественные параметры, признаки и случаи и т.д.
Большинство NER классификаторов базируются на алгоритме CRF (Conditional random field), который относится к классу скрытых Марковских моделей https://nlp.stanford.edu/software/CRF-NER.shtml
Для создания моделей NER классификации мы используем фреймворк с открытым исходным кодом для обработки естественного языка (NLP) - «SpaCy». Он написан на Python, выполняет токенизацию, разметку части речи (PoS) и разбор зависимостей.
Как показало исследование Choi et al, Spacy – это самый быстрый из доступных анализаторов NLP, обеспечивающий высокую точность извлечения признаков. В работе https://www.aclweb.org/anthology/P15-1038.pdf оценивались 10 различных готовых систем извлечения признаков на точность и скорость извлечения. В итоге SpaCy показал лучшую скорость извлечения, поддерживая сопоставимую точность от 85% до 90%.
Модели SpaCy предсталяют собой сверточные нейросети (CNN), которые позволяют делать прогноз, основаннный на представленных во время обучения примерах. Для обучения модели Spacy нужны тексты с размеченными метками именованных сущностей, которые модель должна предсказывать. Дополнительная информация тут: https://spacy.io/.
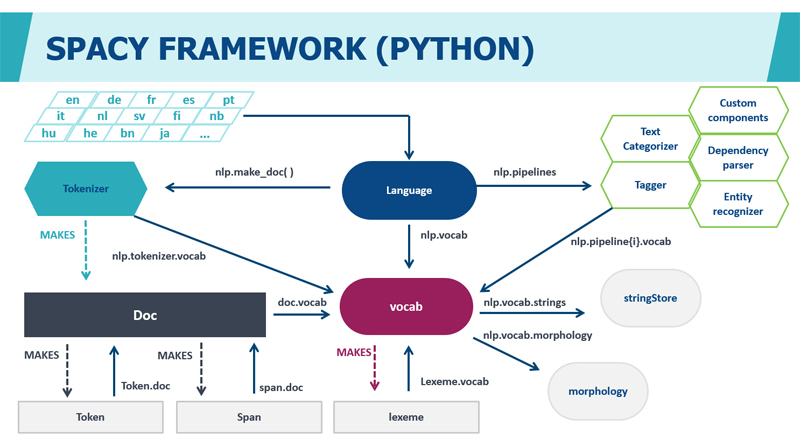
Помимо Spacy при разработке моделей машинного обучения используются пакеты и библиотеки: Keras, OpenNN, PaddlePaddle, PyTorch, TensorFlow, Theano, Torch.
Обучение моделей
Для создания моделей мы накапливаем разнообразные российские данные из электронных медицинских карт с различными заболеваниями: сердечно-сосудистыми заболеваниями, пневмониями, гастритами, ОРВИ, ХОБЛ и др. Обучение моделей NER происходит на основе деперсонифицированных данных из ЭМК, преимущественно это медицинские записи пациентов в возрасте от 30 до 80 лет, которые содержат неструктурированный текст из записей врачей при первичном осмотре, в том числе это объективные данные (рост, вес, АД, частота дыхания), жалобы пациента (повышенная температура, одышка, кашель, изжога), а также признаки лабораторных данных (глюкоза, холестерин, креатинин, мочевина). Для извлечения каждого медицинского текста при обучении используется порядка 200 -500 размеченных текстов с различными написаниями врачами.
Метрики моделей
Для оценки качества моделей используются различные алгоритмы оценки, а для задач NER как правило, используются метрики:
• Precision - точность
• Recall - полнота
• F1 - среднее гармоническое точности и полноты
В связи с успешным применением данного подхода для излечения любой информации была достигнута договоренность, что SOTA NER (наилучшее значение метрик) считается, когда F1 больше 0,9. В наших моделях F-мера составляет порядка 0,986.
Разработанные модели извлечения признаков из медицинских записей
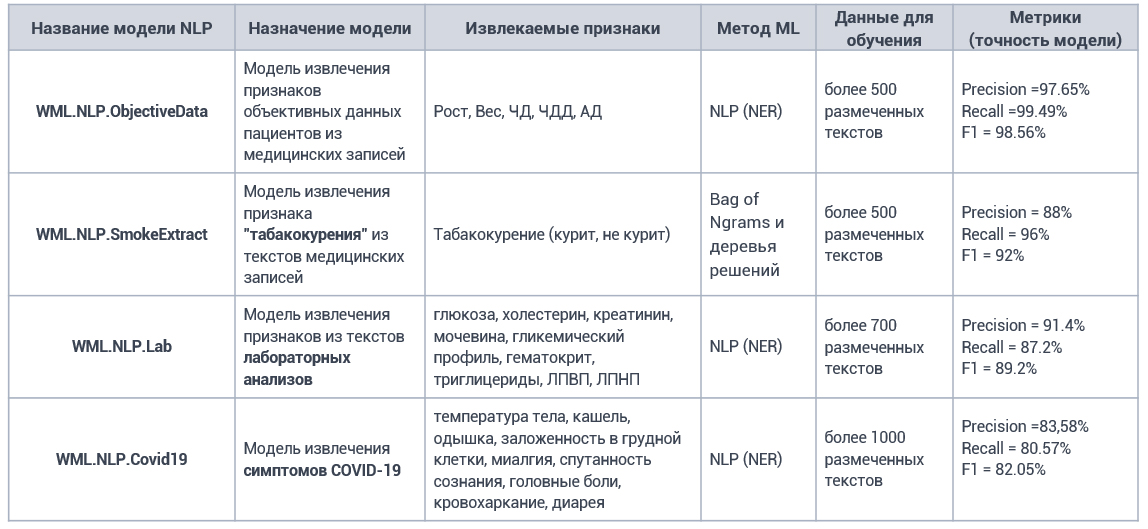
Пример работы сервиса извлечения признаков из медицинских текстов в Webiomed:
Рассмотрим один из примеров врачебной записи:
Жалобы на малопродуктивный кашель, насморк, першение в горле, головные боли, общую слабость, Т 37,5. Около 1 недели назад были боли в животе, однократная рвота, вызвыали СМП. На момент вызова болей нет, беспокоят ежедневный жидкий стул, мацерация ануса. OBJECTIVE: Вес-101, рост- 160 см., АД 140/100, Курит 5 лет. Кожа чистая. Периферические лимфоузлы не увеличены. Щитовидная железа 0 ст. Дыхание везикулярное, без хрипов. ЧД- 18. ЧСС 70 в мин., ритмичный. Б/х крови 27.09.17г.: Общ.белок 72.3 г/л, Билирубин общ. 12.4 мкмоль/л, пр. - 2.4 мкмоль/л, АЛТ 23.8 Е/л, АСТ 34.0 Е/л, Глюкоза крови 5.3 ммоль/л, Мочевина 5.4 ммоль/л, Креатинин 112.3 мкмоль/л, Холестерин 6.7 ммоль/л, Триглицериды 1.60 ммоль/л, РФ - отр., СРБ 36 мг/л, ЛПВП 1.25 ммоль/л, ЛПНП 4.73 ммоль/л, ПТИ 103.1%. МНО 0.96.
Результат извлечения признаков:
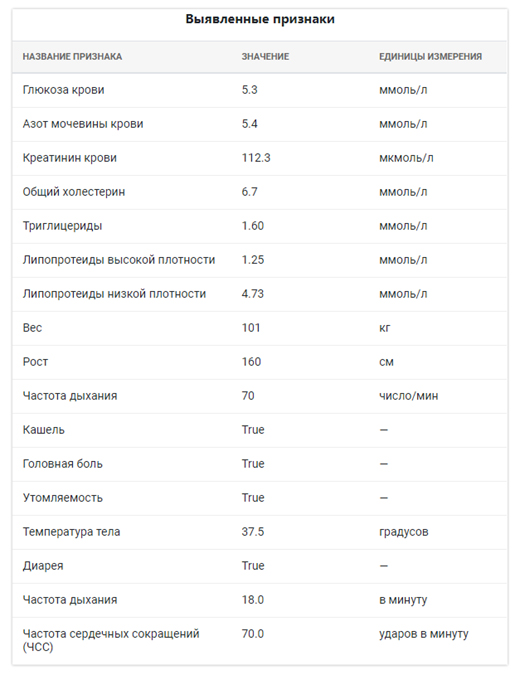
Выводы:
- Разработанные компанией модели извлечения признаков из медицинских текстовых записей методами NLP показали высокую точность при обработке неструктурированной ЭМК пациента. На данный момент они проходят комплексное тестирование на реальных данных пилотных проектов медицинских организаций для выявления предикторов развития заболеваний.
- Планируется значительное расширение числа извлекаемых признаков моделями NLP для дальнейшего развития предиктивных способностей нашей системы для различных заболеваний и факторов риска пациентов.
- Метод извлечения признаков позволяет использовать очень большое количество данных из ЭМК для машинного обучения. Речь идет о сотнях тысячах записей, что практически невозможно реализовать в клинических исследованиях, таких как Framingham Heart Study, где было обследовано около 10 тысяч пациентов за длительный период.
- Сервис Webiomed.NLP может быть интегрирован в любую медицинскую информационную систему для повышения ее функциональных возможностей по интерпретации врачебных записей в ЭМК.
Результаты нашей работы были представлены в публикации: